
Utiliser l'Application de Standardisation des Noms Taxonomiques
taxonomic-app-fr.RmdIntroduction
La fonction launch_taxonomic_match_app() fournit une
application Shiny interactive pour standardiser les noms taxonomiques
contre la base de données taxonomique de référence des plantes d’Afrique
centrale. Cette interface visuelle est idéale pour :
- Explorer et nettoyer les données taxonomiques de manière interactive
- Comprendre la qualité des correspondances grâce au retour visuel
- Examiner manuellement les correspondances incertaines
- Enrichir les données avec des traits au niveau de l’espèce
Prérequis
Avant de lancer l’application, assurez-vous d’avoir :
-
Des identifiants de base de données configurés
(voir
setup_db_credentials()) -
Des données à standardiser dans l’un de ces formats
:
- Fichier Excel (.xlsx, .xls)
- Fichier CSV (.csv)
- Une colonne contenant des noms taxonomiques (ex. genre + espèce) ou des colonnes séparées pour le genre, l’espèce et la famille
Démarrage Rapide
Lancez l’application avec une seule commande :
Alternativement, pré-chargez vos données :
# Avec un data.frame R
my_data <- read.csv("inventaire_arbres.csv")
launch_taxonomic_match_app(data = my_data, name_column = "nom_espece")
# Ajuster la sensibilité de la correspondance approximative (par défaut 0.7)
launch_taxonomic_match_app(min_similarity = 0.5) # Correspondance plus permissiveGuide Étape par Étape
Phase 1 : Vue Initiale
Lorsque vous lancez l’application pour la première fois, vous verrez l’interface principale avec une barre latérale pour la configuration et des onglets pour les différentes phases du flux de travail :
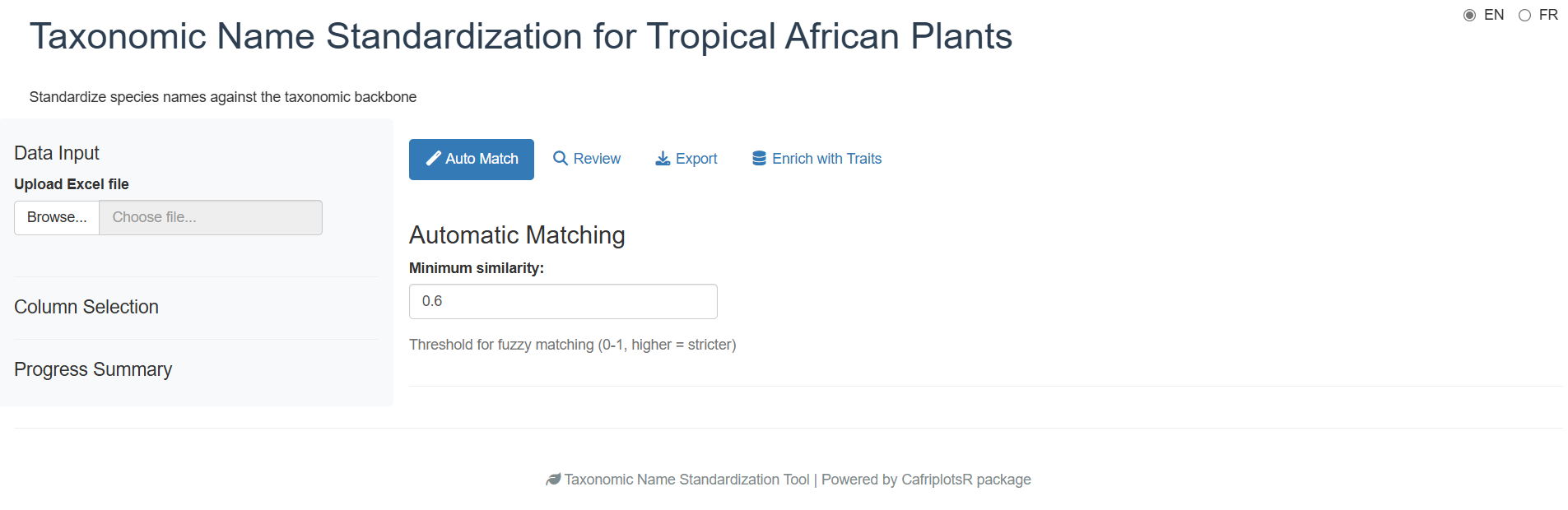
L’application utilise un flux de travail par onglets qui vous guide à travers chaque phase séquentiellement :
- Auto Match - Correspondance automatique
- Review - Révision manuelle des noms non appariés
- Export - Téléchargement des résultats
- Traits Enrichment - Ajout de traits d’espèces
Phase 2 : Charger Vos Données
La première étape est de fournir vos données. L’application offre deux méthodes d’import :
Import par Fichier (Par Défaut)
- Charger un fichier Excel en utilisant le navigateur de fichiers (supporte .xlsx, .xls)
- Charger un fichier CSV
-
Utiliser des données R pré-chargées (si vous avez
passé le paramètre
data)
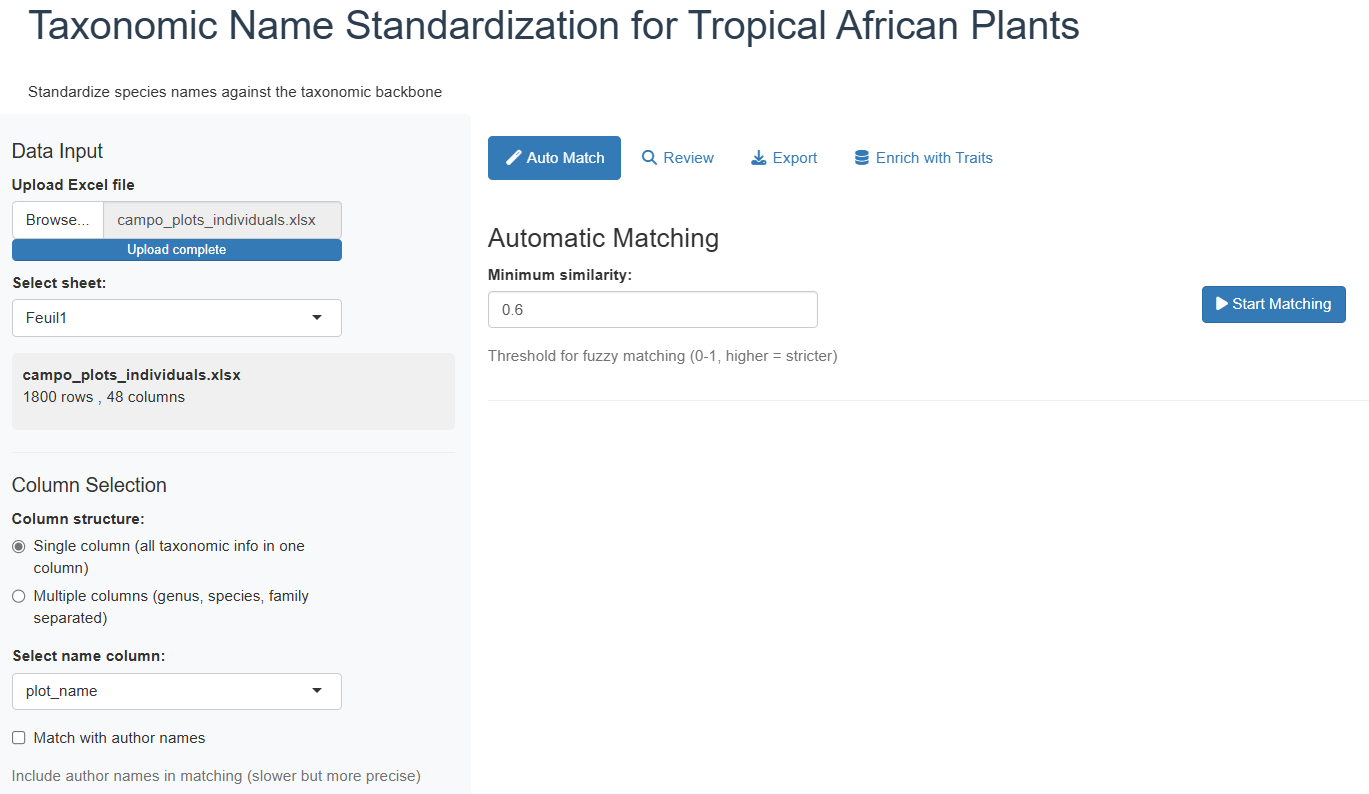
Pour les fichiers Excel avec plusieurs feuilles, vous pouvez sélectionner quelle feuille utiliser. L’application affichera un aperçu de vos données chargées pour que vous puissiez vérifier qu’elles ont été lues correctement.
Saisie Texte (Copier-Coller) - NOUVEAU
Pour standardiser rapidement quelques noms, ou lorsque vous avez une liste copiée depuis une autre source, utilisez la méthode Saisie texte :
- Sélectionnez “Texte (coller/saisir)” dans les boutons radio de méthode d’import
- Collez ou tapez vos noms taxonomiques dans la zone de texte
- Cliquez sur “Charger les noms” pour traiter la saisie
Séparateurs acceptés : - Un nom par ligne
(recommandé) - Séparés par virgule :
Lophira alata, Terminalia superba, Aucoumea klaineana -
Séparés par point-virgule :
Lophira alata; Terminalia superba; Aucoumea klaineana -
Séparés par tabulation (utile lors du copier-coller depuis Excel)
L’application automatiquement : - Supprime les lignes vides et les
espaces superflus - Supprime les doublons (en préservant l’ordre) - Crée
une colonne unique nommée taxon_name pour l’appariement
Cette méthode est idéale pour : - Vérifier rapidement quelques noms d’espèces - Coller des listes depuis des emails ou documents - Tester l’application sans préparer de fichier
Phase 3 : Sélectionner la/les Colonne(s) de Noms
Une fois les données chargées, vous avez deux options pour sélectionner les noms taxonomiques :
Mode Colonne Unique (Par Défaut)
Sélectionnez une colonne contenant le nom taxonomique complet :
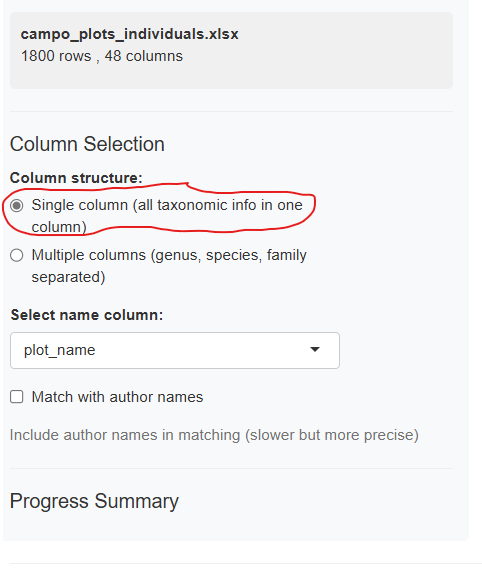
Le menu déroulant affiche toutes les colonnes disponibles de votre jeu de données. Choisissez celle contenant les noms d’espèces (généralement formatés comme “Genre espèce” ou “Genre espèce Auteur”).
Mode Colonnes Multiples (NOUVEAU)
Si vos données ont des colonnes séparées pour le genre, l’espèce et la famille, activez “Utiliser plusieurs colonnes” :
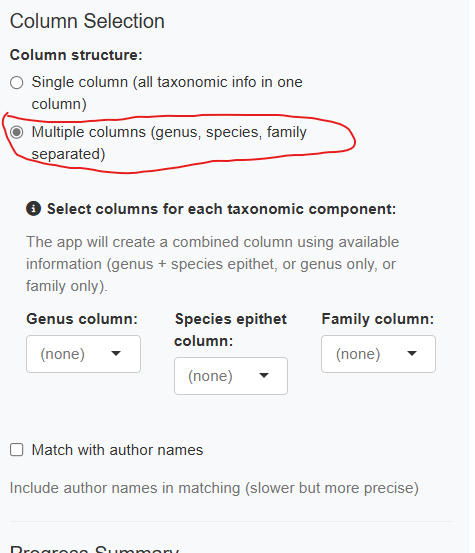
L’application combinera automatiquement ces colonnes en un seul nom taxonomique pour la correspondance, en utilisant une approche hiérarchique : - Si genre et espèce sont disponibles : “Genre espèce” - Si seulement genre : “Genre” - Si seulement famille : “Famille”
Vous pouvez aussi optionnellement inclure une colonne d’auteur.
Phase 4 : Correspondance Automatique
Cliquez sur le bouton “Démarrer la Correspondance” pour commencer le processus de correspondance automatique. L’application utilise une stratégie de correspondance à cinq niveaux :
- Correspondance exacte sur l’espèce : Recherche directe du nom complet (genre + espèce)
- Correspondance exacte sur le genre : Correspondance au niveau du genre
- Correspondance exacte sur la famille : Correspondance au niveau de la famille
- Correspondance exacte sur la classe : Correspondance à un niveau taxonomique supérieur
- Correspondance approximative : Correspondance de chaînes approximative pour les noms restants
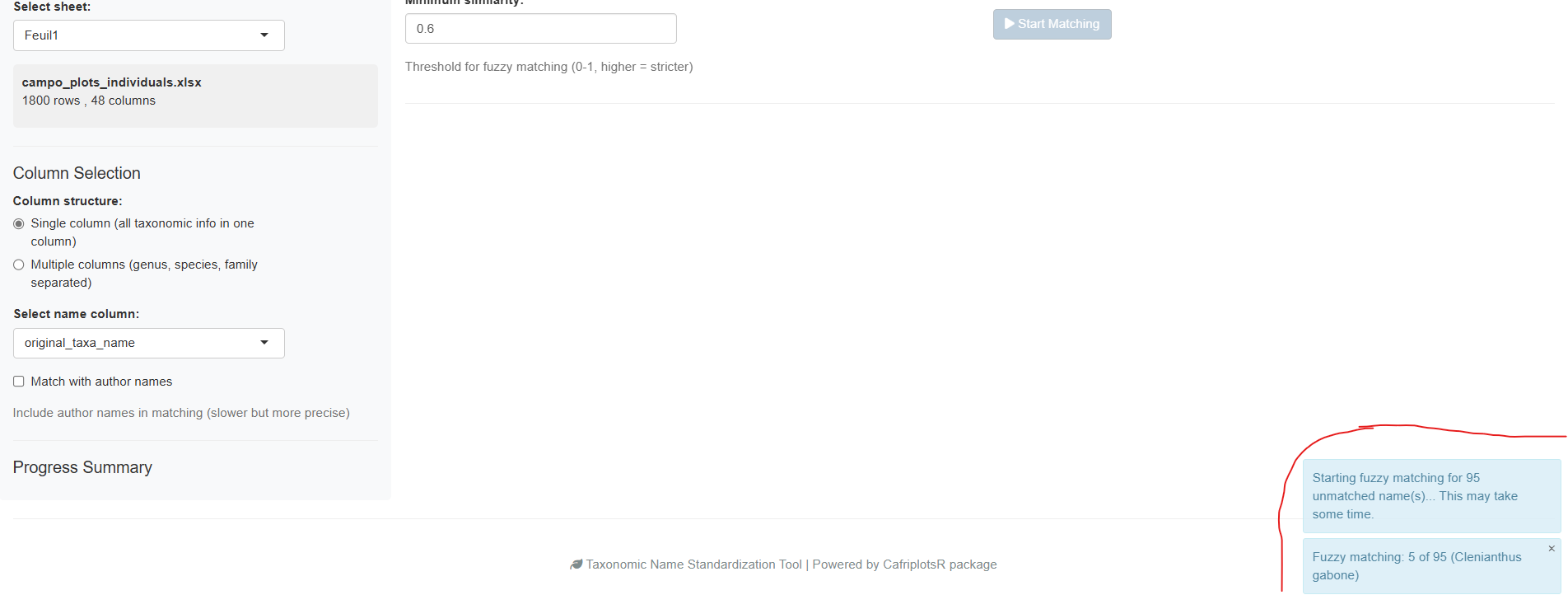
La barre de progression affiche le statut en temps réel. Pour les grands jeux de données, cela peut prendre quelques minutes. La barre latérale affiche des statistiques en direct :
- Nombre de correspondances exactes
- Nombre de correspondances au niveau du genre
- Nombre de correspondances approximatives
- Nombre de noms non appariés
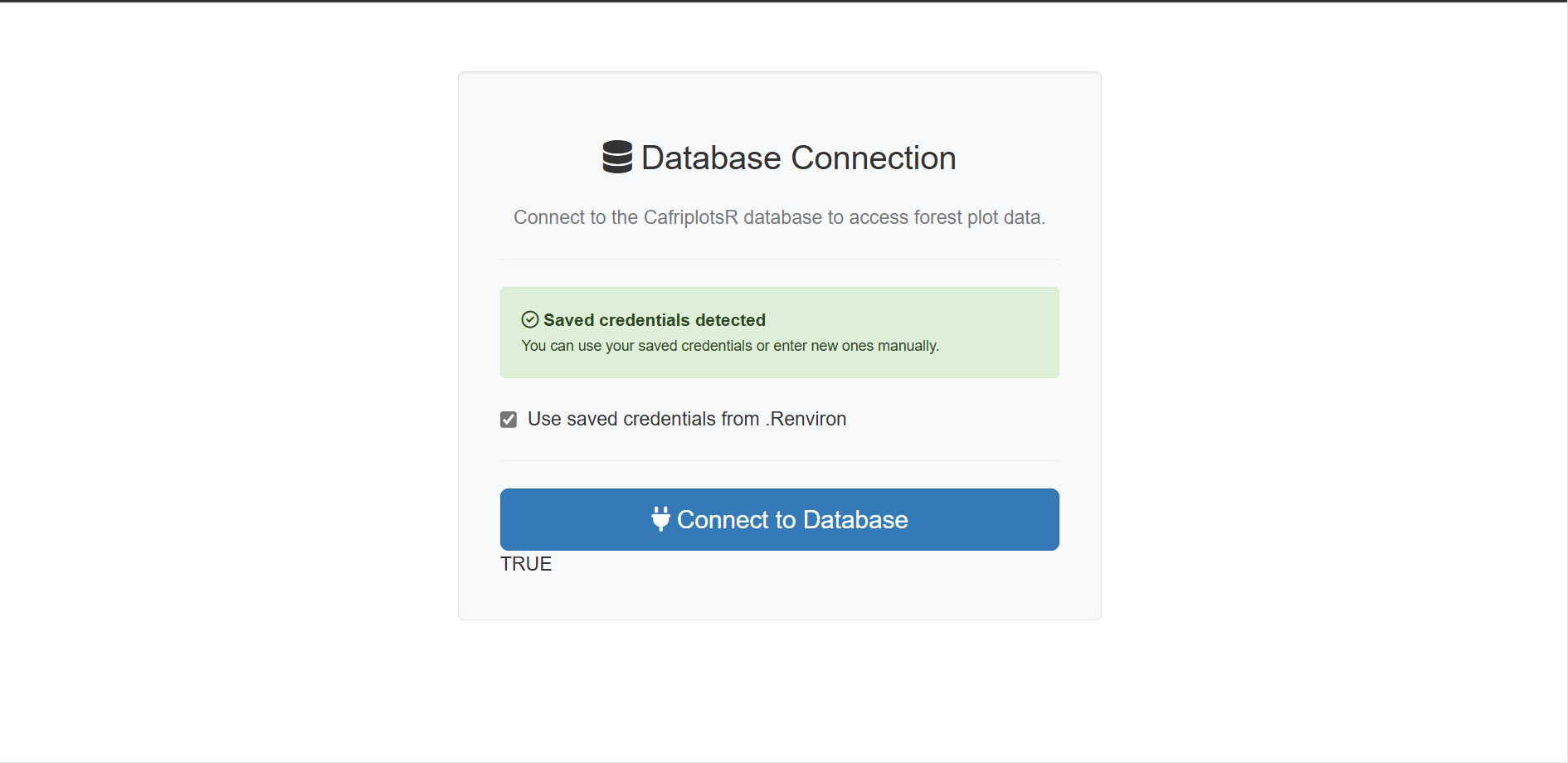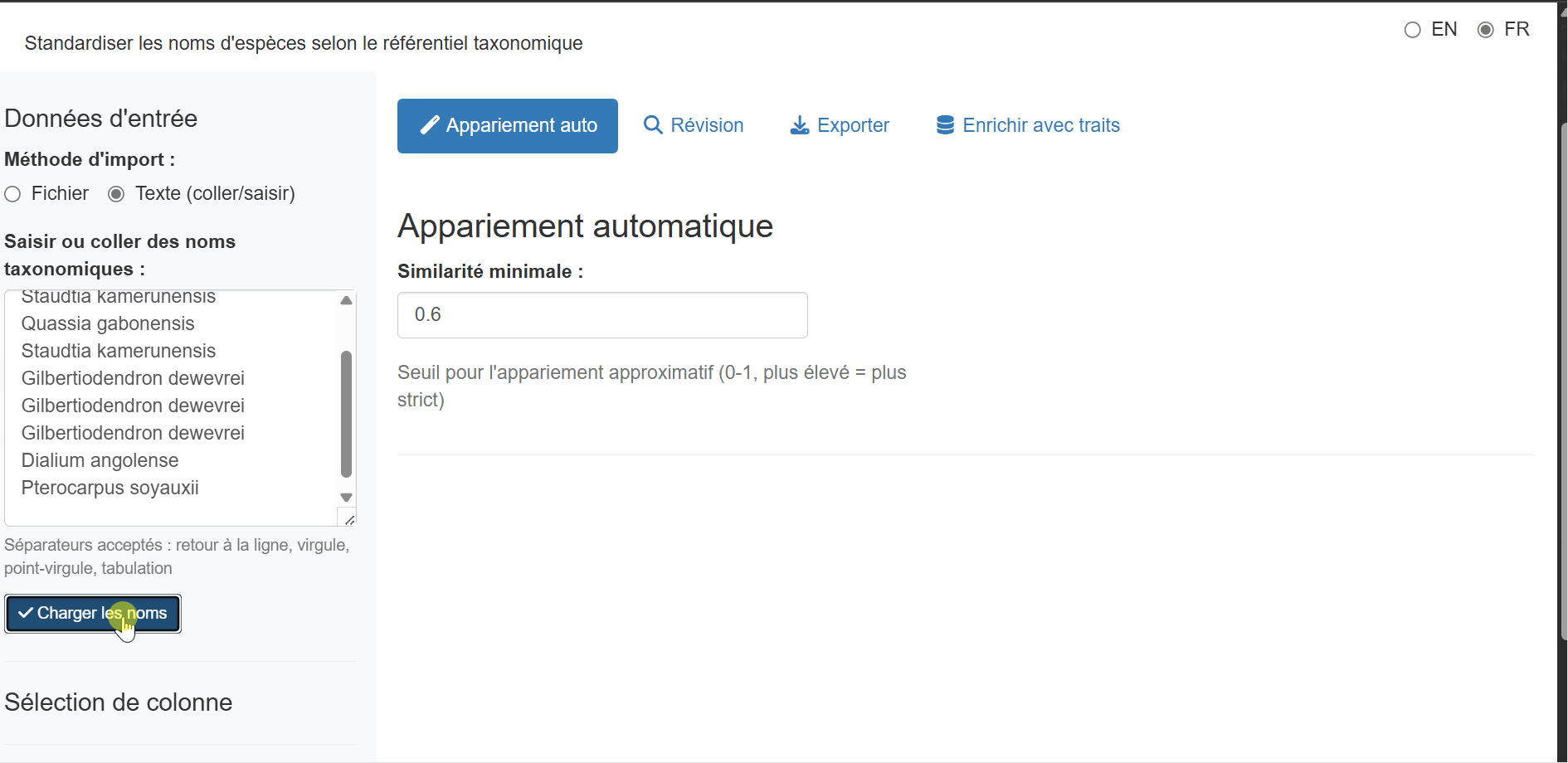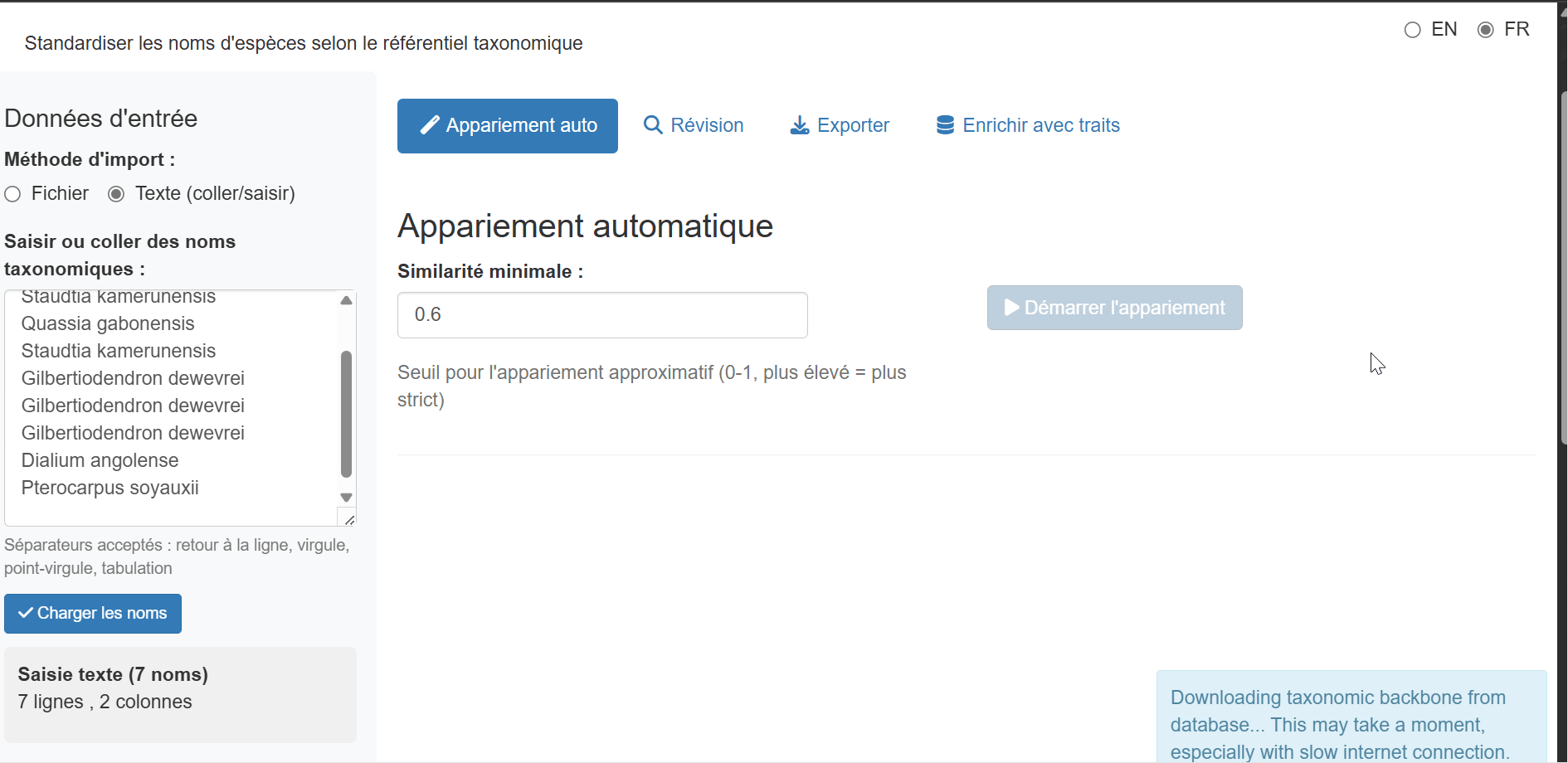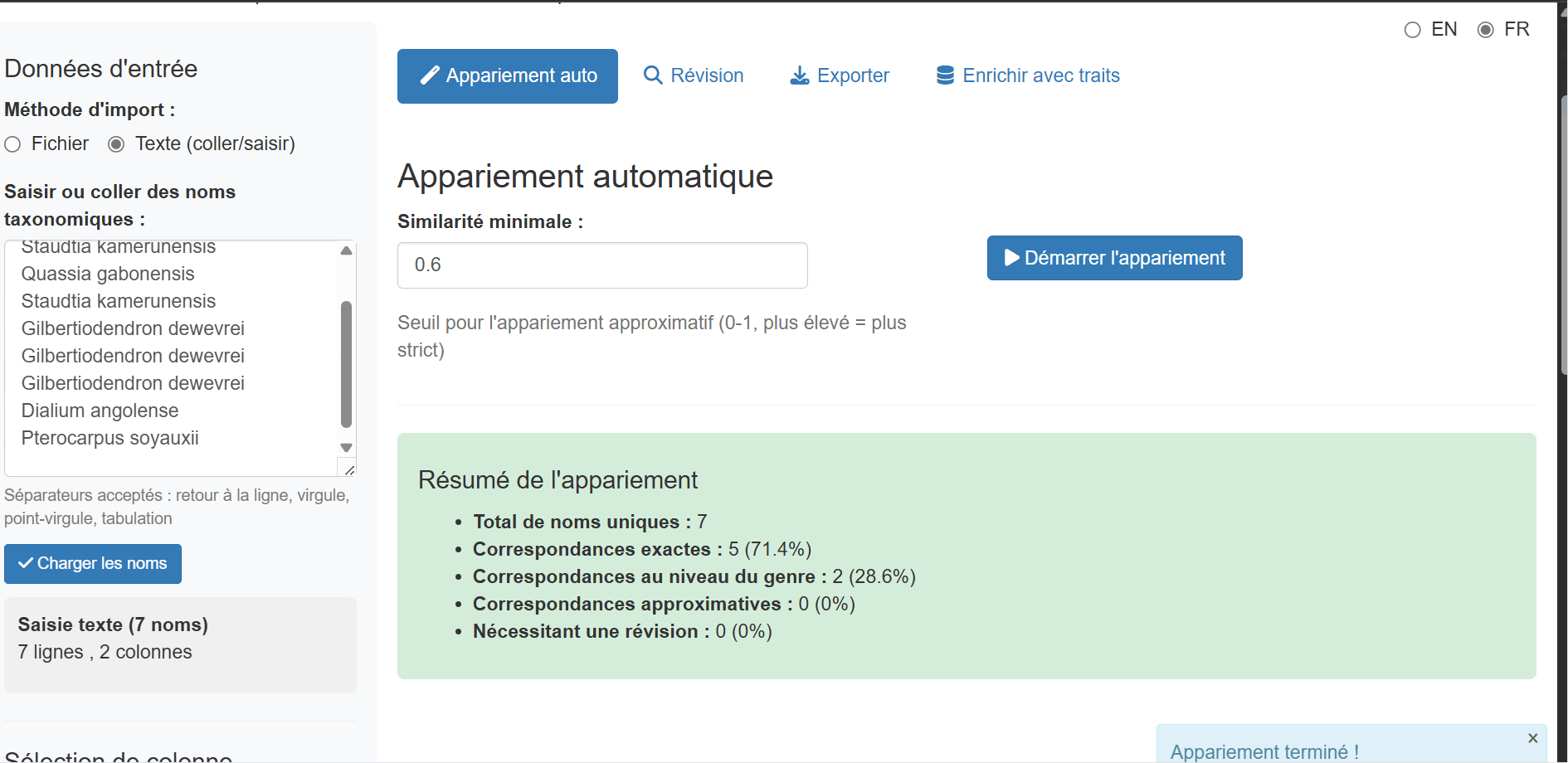
Phase 5 : Examiner les Résultats de Correspondance
Après la fin de la correspondance, l’onglet Auto Match affiche un tableau résumé avec tous les noms et leur statut de correspondance :
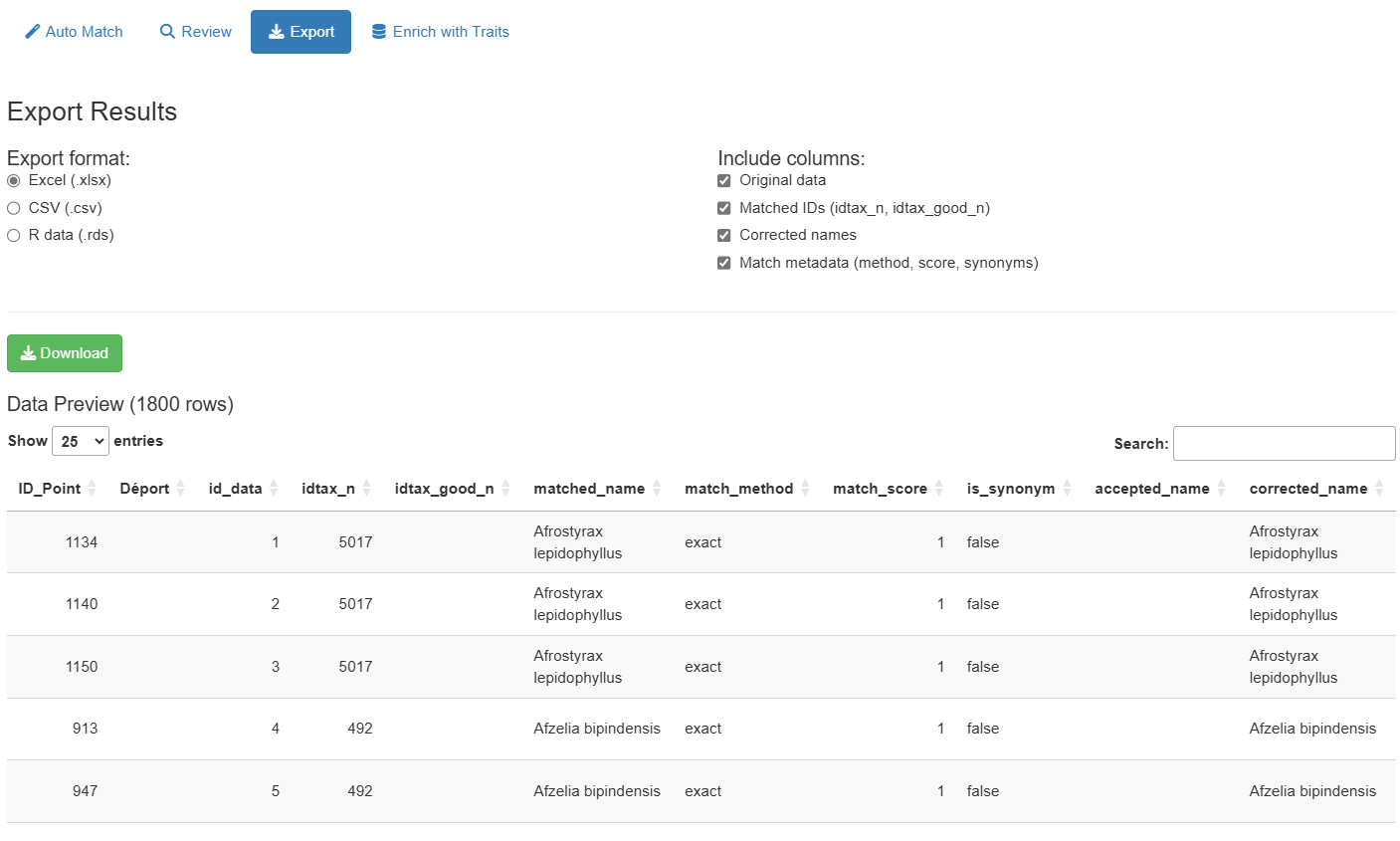
Le tableau des résultats inclut :
- Nom original : Votre nom en entrée
- matched_name : Nom trouvé dans la référence
- match_method : Comment il a été apparié (exact_species, exact_genus, exact_family, fuzzy, manual)
- match_score : Score de similarité (0-1, plus élevé est meilleur)
- idtax_n : ID du taxon dans la base de données
- is_synonym : Si le nom apparié est un synonyme
- accepted_name : Nom accepté actuel (si synonyme)
Indicateurs de qualité de correspondance :
- Correspondance exacte (1.0) : Correspondance parfaite, pas de révision nécessaire
- Haute similarité (>0.8) : Très probablement correct, révision rapide recommandée
- Similarité moyenne (0.5-0.8) : Correspondance possible, révision suggérée
- Basse similarité (<0.5) : Incertain, révision manuelle requise
- Pas de correspondance : Nécessite une sélection manuelle
Phase 6 : Révision Manuelle
Pour les noms non appariés ou incertains, passez à l’onglet “Review” pour réviser manuellement et sélectionner les correspondances :
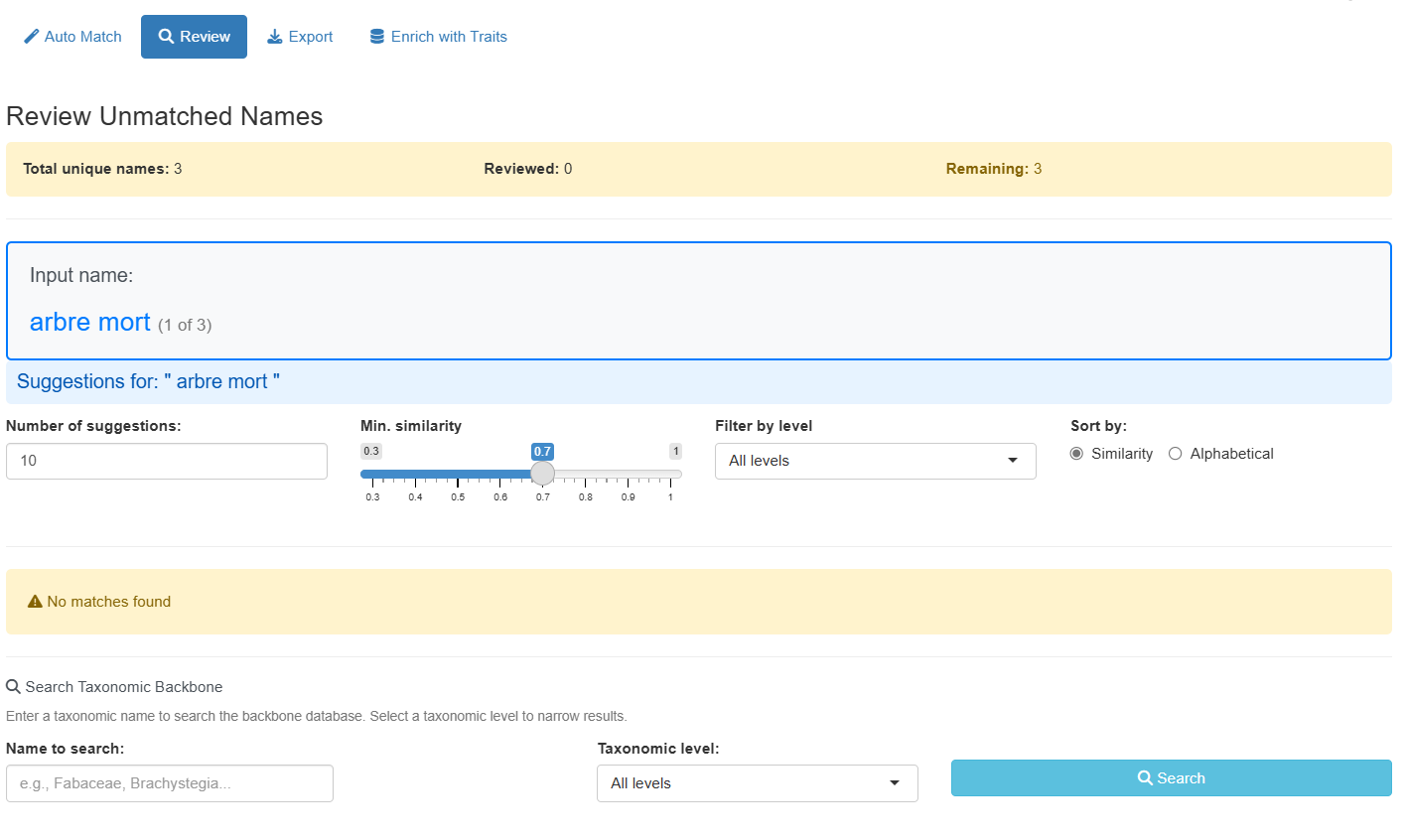
L’interface de révision fournit deux façons de trouver des correspondances :
Panneau de Suggestions Approximatives
Affiche des suggestions automatiques classées par similarité avec des options de filtrage avancées :
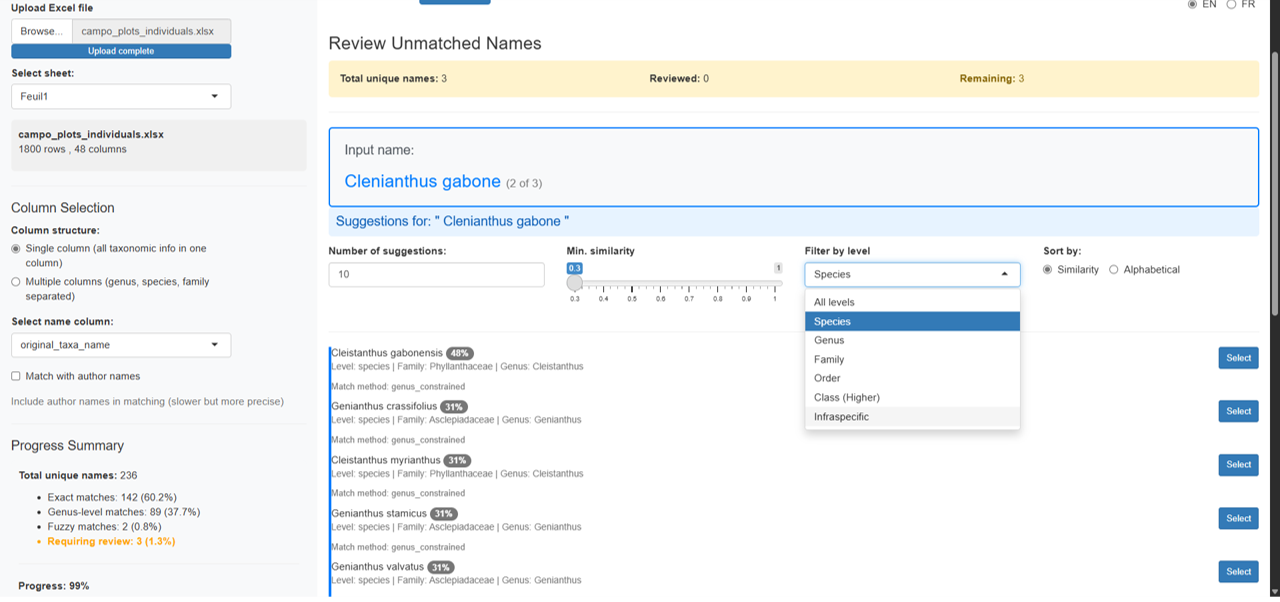
Options de filtrage :
- Nombre de suggestions : Curseur pour afficher 5-30 suggestions
- Similarité minimale : Ajuster le seuil (0.3-1.0)
- Filtre de niveau taxonomique : Filtrer par Tous, Espèce, Genre, Famille, Ordre, Classe ou Infraspécifique
- Trier par : Score de similarité ou ordre alphabétique
Chaque carte de suggestion affiche :
- Nom avec badge de similarité coloré (vert = élevé, bleu = moyen, jaune = bas)
- Niveau taxonomique et famille
- Information de synonymie si applicable
- Bouton Sélectionner pour acceptation en un clic
Panneau de Recherche Manuelle
Pour les noms sans bonnes suggestions, utilisez la recherche manuelle :
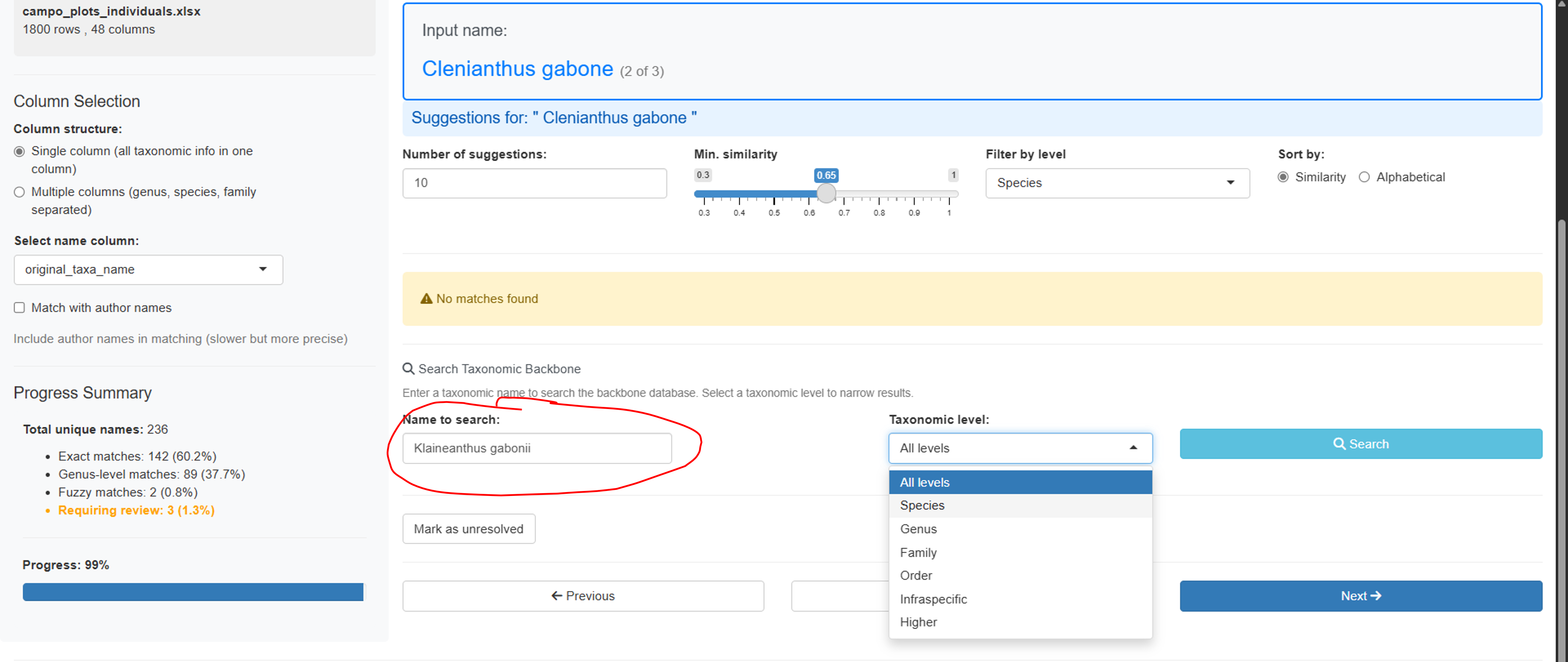
- Tapez n’importe quel terme de recherche pour interroger la référence taxonomique
- Filtrez les résultats par niveau taxonomique
- Voir les informations détaillées pour chaque correspondance
- Sélectionnez la bonne correspondance ou marquez comme “non résolu”
Navigation :
- Utilisez les boutons Précédent/Passer/Suivant pour parcourir les noms non appariés
- Le compteur de progression affiche les noms révisés vs. restants
- L’application mémorise vos sélections et met à jour automatiquement les résultats
Phase 7 : Enrichir les Données avec des Traits
Passez à l’onglet “Traits Enrichment” pour ajouter des traits au niveau de l’espèce à vos données appariées :
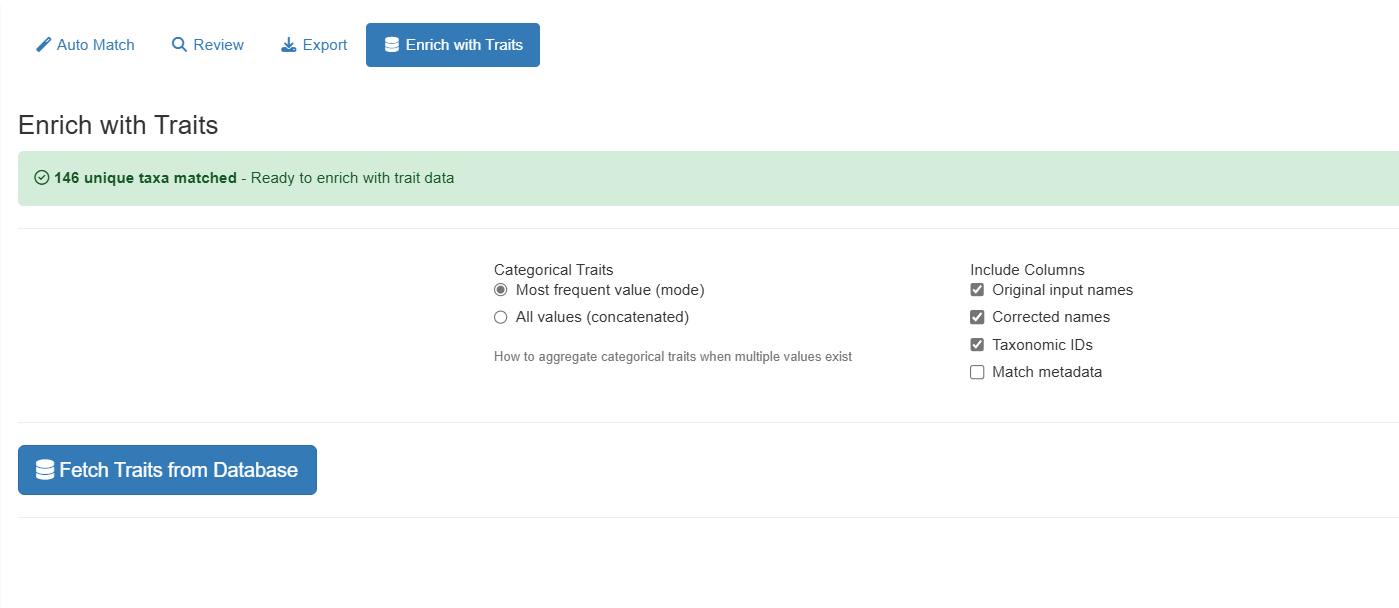
Options :
-
Mode d’agrégation catégorielle :
- “mode” - Utiliser la valeur la plus fréquente par taxon
- “concat” - Concaténer toutes les valeurs uniques
-
Sélectionner les colonnes à inclure :
- Noms d’entrée originaux
- Noms corrigés
- IDs taxonomiques
- Métadonnées de correspondance
Les traits disponibles incluent :
- Forme de croissance
- Densité du bois
- Traits foliaires
- Caractéristiques écologiques
Les données enrichies combinent vos taxons appariés avec les traits sélectionnés :
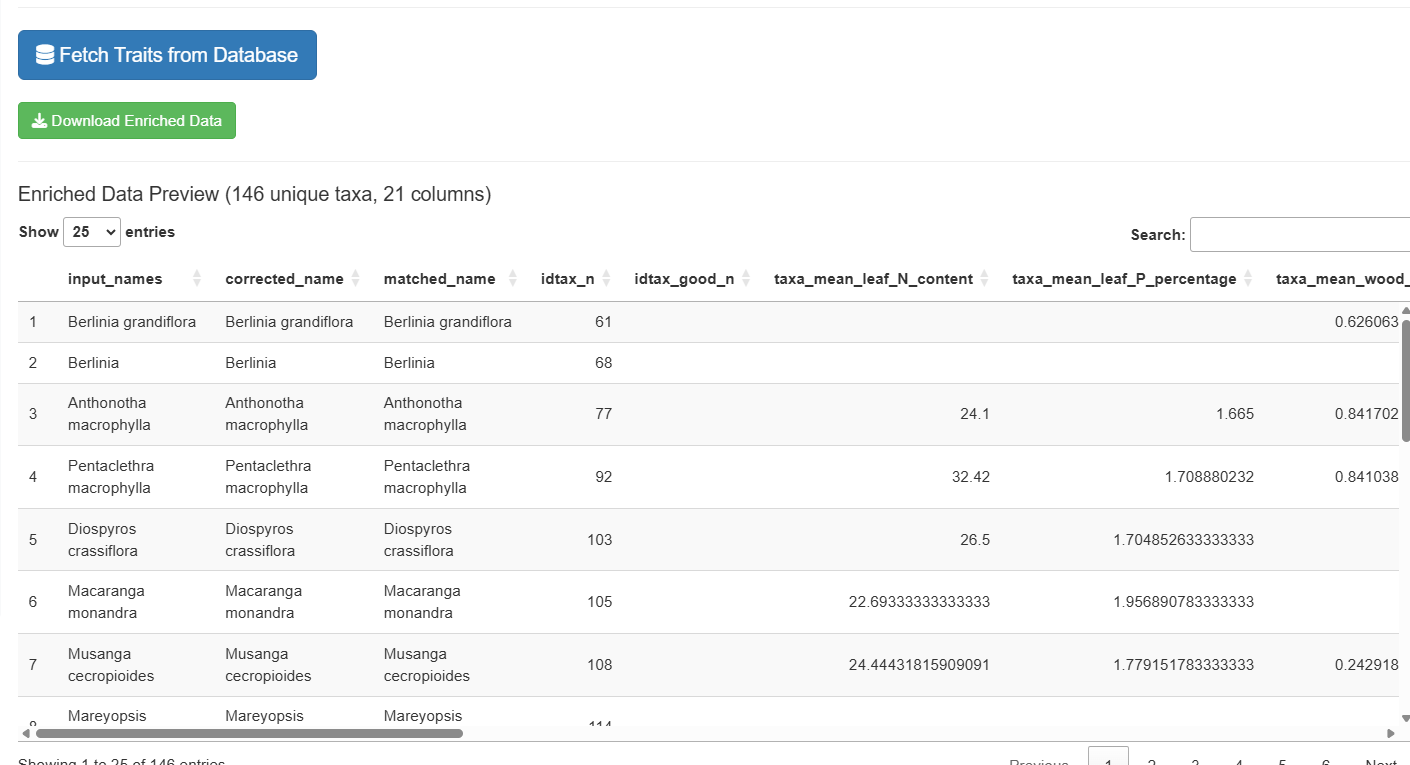
Note : L’export enrichi crée une ligne par taxon unique, pas par ligne d’entrée. Les noms d’entrée sont concaténés avec des séparateurs pipe.
Phase 8 : Exporter les Résultats
Passez à l’onglet “Export” pour télécharger votre jeu de données standardisé :
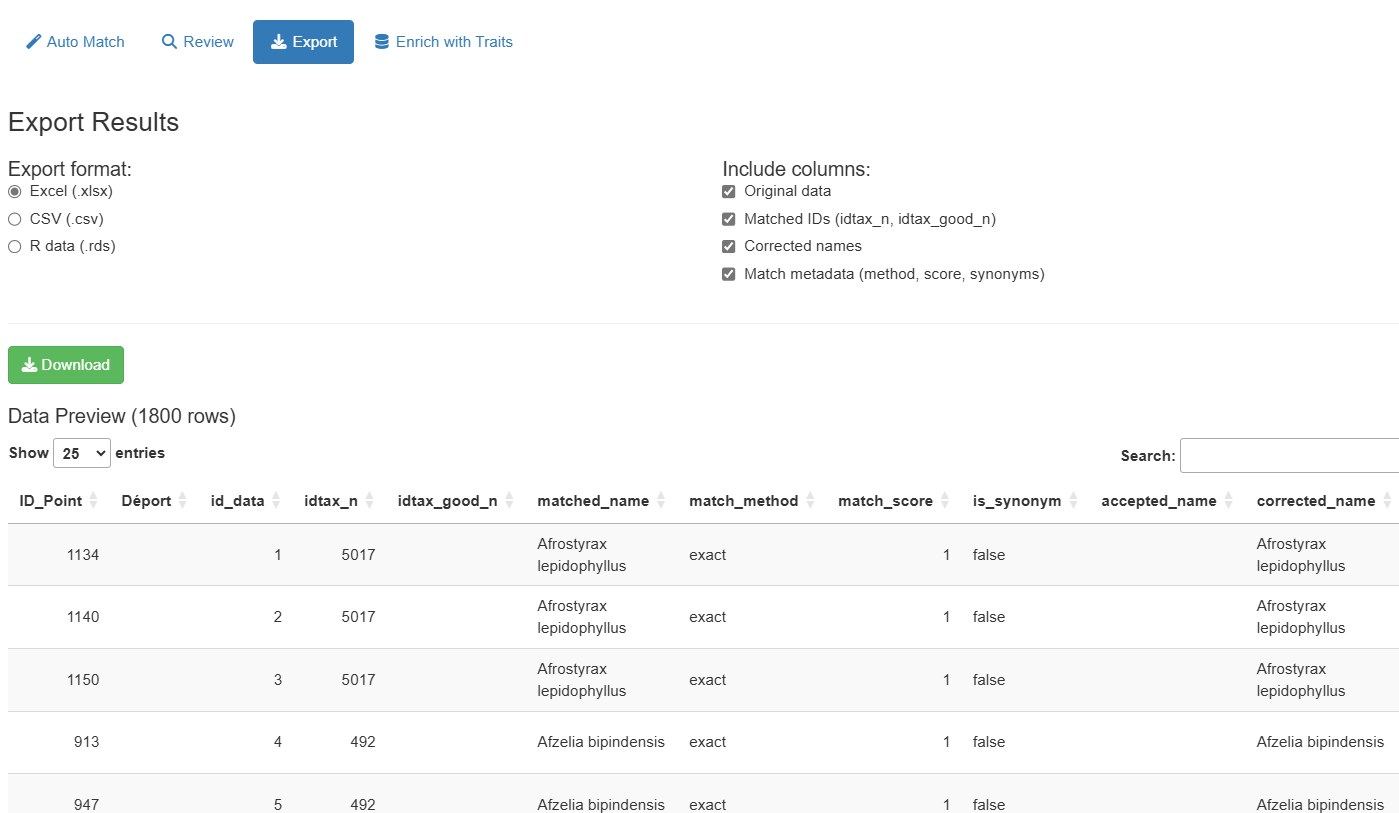
Formats disponibles :
- Excel (.xlsx) : Idéal pour partager avec des collaborateurs
- CSV (.csv) : Format tabulaire universel
- RDS (.rds) : Format natif R préservant les types de données
Colonnes sélectionnables :
- Données originales (toutes vos colonnes d’entrée)
- IDs appariés (idtax_n, idtax_good_n)
- Noms corrigés (corrected_name, matched_name)
- Métadonnées de correspondance (match_method, match_score, is_synonym, accepted_name)
Un tableau de prévisualisation montre les données avant l’export avec des contrôles de pagination.
Comprendre les Colonnes de Sortie
L’application ajoute ces colonnes à vos données :
| Colonne | Description |
|---|---|
idtax_n |
ID du taxon apparié dans la base de données de référence |
idtax_good_n |
ID du taxon accepté (pour les synonymes) |
matched_name |
Nom trouvé dans la référence |
corrected_name |
Nom standardisé final |
match_method |
Stratégie de correspondance utilisée (exact_species, exact_genus, exact_family, fuzzy, manual, unresolved) |
match_score |
Score de similarité (0-1) |
is_synonym |
TRUE si le nom apparié est un synonyme |
accepted_name |
Nom accepté actuel (si synonyme) |
family |
Famille taxonomique |
genus |
Genre taxonomique |
Options Avancées
Sélection de la Langue
L’application supporte désormais l’opération bilingue avec des interfaces en français et en anglais. Le français est la langue par défaut.
Dans l’Interface de l’Application:
Un sélecteur de langue est situé en haut à droite de l’application : - Cliquez sur “FR” pour l’interface en français - Cliquez sur “EN” pour l’interface en anglais
Le changement de langue est instantané et affecte tous les éléments de l’interface incluant : - Les libellés des onglets - Le texte des boutons - Les instructions et textes d’aide - Les en-têtes de colonnes - Les messages d’erreur et notifications
Définir la Langue Initiale par Programme:
# Lancer l'application en anglais
launch_taxonomic_match_app(language = "en")
# Lancer l'application en français (par défaut)
launch_taxonomic_match_app(language = "fr")
# ou simplement :
launch_taxonomic_match_app()Le paramètre de langue est interactif - les utilisateurs peuvent changer de langue à tout moment pendant leur session sans perdre leur progression ou leurs données.
Ajuster la Correspondance Approximative
Contrôlez la sensibilité de la correspondance avec le paramètre
min_similarity :
# Très strict - uniquement des correspondances de haute qualité
launch_taxonomic_match_app(min_similarity = 0.8)
# Paramètre par défaut
launch_taxonomic_match_app(min_similarity = 0.7)
# Plus permissif - permet des correspondances de moindre qualité
launch_taxonomic_match_app(min_similarity = 0.5)Des valeurs plus basses ratissent plus large mais peuvent inclure des faux positifs. Des valeurs plus élevées sont plus conservatrices mais peuvent manquer des correspondances valides.
Augmenter les Suggestions
Afficher plus de suggestions de correspondance approximative par nom :
# Afficher les 20 meilleures suggestions au lieu des 10 par défaut
launch_taxonomic_match_app(max_suggestions = 20)Utile quand les suggestions initiales n’incluent pas la bonne correspondance. Vous pouvez aussi ajuster ceci de manière interactive dans l’onglet Review en utilisant le curseur.
Paramètres de la Fonction
launch_taxonomic_match_app(
data = NULL, # Optionnel : pré-charger un data.frame
name_column = NULL, # Optionnel : pré-sélectionner une colonne
min_similarity = 0.7, # Seuil de correspondance approximative (0-1)
max_suggestions = 10 # Max suggestions par nom non apparié
)Dépannage
Problèmes de Connexion
Problème : “Échec de connexion à la base de données”
Solution :
# Vérifier la connexion
db_diagnostic()
# Réinitialiser les identifiants si nécessaire
remove_db_credentials()
setup_db_credentials()Pas de Correspondances Approximatives Trouvées
Problème : Aucune suggestion n’apparaît pour les noms non appariés
Causes possibles : - Seuil
min_similarity trop élevé - Les noms taxonomiques
contiennent des fautes de frappe ou un formatage non standard - Les noms
ne sont pas présents dans la référence taxonomique (ex. taxons non
africains)
Solutions : - Diminuer min_similarity :
launch_taxonomic_match_app(min_similarity = 0.5) - Utiliser
le filtre de niveau taxonomique pour chercher au niveau du genre ou de
la famille - Nettoyer les noms en entrée (supprimer les espaces
supplémentaires, corriger les fautes évidentes) - Vérifier que les noms
sont des taxons africains
Performance de Correspondance Lente
Problème : La correspondance prend très longtemps pour les grands jeux de données
Solutions : - Utiliser le traitement par lots à la
place : match_taxonomic_names() pour un flux de travail
programmatique - Traiter les données en morceaux (diviser les grands
jeux de données) - L’application télécharge toute la référence une fois
pour l’efficacité, donc le chargement initial peut être lent
Quand Utiliser l’Application vs. l’Approche Programmatique
Utilisez l’Application Shiny quand :
- Exploration interactive des données
- Vous préférez les interfaces visuelles
- Le jeu de données est de petite à moyenne taille (<5 000 lignes)
- Besoin de réviser manuellement les correspondances incertaines
- Apprentissage du processus de correspondance
Utilisez match_taxonomic_names() quand :
- Traitement de grands jeux de données (>5 000 lignes)
- Automatisation des flux de travail dans des scripts
- Intégration avec des pipelines de données
- La reproductibilité est critique (NE JAMAIS SUPPRIMER LA COLONNE CONTENANT LE NOM ORIGINAL)
- Traitement par lots de plusieurs fichiers
Exemple d’approche programmatique :
# Charger les données
my_data <- read.csv("inventaire_arbres.csv")
# Apparier les noms
matched <- match_taxonomic_names(
names = my_data$nom_espece,
min_similarity = 0.7
)
# Fusionner avec les données originales
result <- cbind(my_data, matched)
# Exporter
write.csv(result, "inventaire_standardise.csv", row.names = FALSE)Voir Aussi
-
match_taxonomic_names(): Fonction de correspondance sous-jacente pour usage programmatique -
query_taxa(): Interroger directement la référence taxonomique -
match_tax(): Fonction simple de recherche taxonomique -
vignette("using-query-plots-fr"): Guide pour interroger les données de parcelles
Conseils pour de Meilleurs Résultats
- Nettoyez d’abord vos données : Supprimez les fautes de frappe évidentes, les espaces supplémentaires et les caractères spéciaux
- Comprenez vos données : Sachez quels groupes taxonomiques sont dans votre jeu de données
- Utilisez le mode multi-colonnes : Si vous avez des colonnes séparées genre/espèce/famille, combinez-les pour une meilleure correspondance
- Filtrez par niveau taxonomique : Utilisez le filtre de niveau dans l’onglet Review pour trouver des correspondances au genre ou à la famille
- Examinez les scores de correspondance : N’acceptez pas aveuglément les correspondances à faible similarité (<0.6)
- Sauvegardez de manière incrémentale : Exportez les résultats intermédiaires pour éviter de perdre le travail de révision manuelle
-
Documentez les paramètres : Notez quelle valeur de
min_similarityvous avez utilisée pour la reproductibilité
Exemple de Flux de Travail
Voici un flux de travail complet du début à la fin :
# 1. Charger vos données
trees <- read.csv("inventaire_forestier.csv")
# Colonnes : plot_id, tree_number, species_name, dbh, height
# 2. Lancer l'application avec les données
launch_taxonomic_match_app(
data = trees,
name_column = "species_name",
min_similarity = 0.7
)
# 3. Dans l'application :
# - Revoir les correspondances automatiques dans l'onglet Auto Match
# - Utiliser l'onglet Review pour résoudre les noms non appariés
# - Appliquer les filtres de niveau taxonomique si nécessaire
# - Optionnellement enrichir avec des traits dans l'onglet Traits Enrichment
# - Exporter comme "inventaire_forestier_standardise.xlsx"
# 4. Continuer l'analyse avec les données standardisées
standardized <- readxl::read_excel("inventaire_forestier_standardise.xlsx")
# Vous avez maintenant des IDs taxonomiques propres pour des analyses ultérieures !Ce flux de travail assure que vos données taxonomiques sont standardisées et prêtes pour des analyses en aval comme les métriques de diversité, les analyses basées sur les traits ou l’intégration dans la base de données.
Captures d’Écran Suggérées
Pour compléter cette documentation, les captures d’écran suivantes devraient être réalisées :
- app-initial-view.png - Interface complète de l’application après lancement avec tous les onglets visibles
- app-upload-data.png - Panneau de chargement des données avec navigateur de fichiers et sélection de feuille
- app-text-input.gif - Interface de saisie texte avec zone de texte et bouton “Charger les noms” (NOUVEAU)
- app-column-select.png - Menu déroulant de sélection de colonne unique
- app-column-select-multi.png - Mode colonnes multiples avec sélecteurs genre/espèce/famille
- app-matching-progress.png - Correspondance en cours avec barre de progression et statistiques en direct
- app-matching-results.png - Tableau des résultats dans l’onglet Auto Match montrant les noms appariés
- app-review-interface.png - Vue d’ensemble de l’onglet Review avec affichage du nom non apparié
- app-review-suggestions.png - Panneau de suggestions approximatives avec options de filtrage (filtre niveau, tri, curseur)
- app-review-manual-search.png - Interface de recherche manuelle avec boîte de recherche et résultats
- app-enrich-data-interface.png - Onglet d’enrichissement des traits avec mode d’agrégation et sélection de colonnes
- app-enrich-data-results.png - Aperçu des données enrichies avec traits
- app-export-options.png - Onglet Export avec sélection de format et cases à cocher des colonnes